Sự khác biệt giữa NGS và Sanger Sequencing | NGS và Sanger Sequencing
Sự khác biệt chính - NGS và Sanger Sequencing
Trình tự thế hệ tiếp theo (NGS) và Sanger Sequencing là hai loại kỹ thuật lập trình trình tự nucleotide được phát triển trong thời gian đó. Phương pháp Sanger Sequencing đã được sử dụng rộng rãi trong nhiều năm và NGS đã thay thế nó gần đây do lợi thế của nó. Sự khác biệt chính giữa NGS và Sanger Sequencing là NGS hoạt động trên nguyên tắc sắp xếp hàng triệu chuỗi đồng thời một cách nhanh chóng thông qua hệ thống trình tự trong khi Sanger Sequencing hoạt động trên nguyên tắc chấm dứt chuỗi do sự kết hợp các didexynucleotides có chọn lọc Enzyme DNA polymerase trong quá trình sao chép DNA và tách rời mảnh bằng điện di mao mạch.
NỘI DUNG
1. Tổng quan và Chênh lệch khác nhau
2. Trình tự nucleotide là gì?
3. NGS là gì
4. Sanger Sequencing là gì
5. So sánh Side by Side - NGS và Sanger Sequencing
6. Tóm tắt
Trình tự nucleotide là gì?
Thông tin di truyền được lưu trữ trong các trình tự nucleotide của DNA hoặc RNA của một sinh vật. Quá trình xác định trật tự chính xác của nucleotide (sử dụng bốn cơ sở) trong một đoạn nhất định (trong một gen, nhóm các gen, nhiễm sắc thể, và bộ gen hoàn chỉnh) được biết đến như trình tự nucleotide . Nó rất quan trọng trong các nghiên cứu về gen, nghiên cứu pháp y, virus học, hệ thống sinh học, chẩn đoán y khoa, công nghệ sinh học và nhiều lĩnh vực khác để phân tích cấu trúc và chức năng của gien. Có nhiều loại phương pháp lập trình khác nhau do các nhà khoa học phát triển. Trong số đó, Sanger trình tự được phát triển bởi Frederick Sanger năm 1977 đã được sử dụng rộng rãi và phổ biến trong một thời gian dài cho đến khi Trình tự thế hệ tiếp theo thay thế nó.
NGS là gì?
Trình tự thế hệ tiếp theo (NGS) là một thuật ngữ được sử dụng để đề cập đến quá trình sắp xếp trình tự thông lượng cao hiện đại. Nó mô tả một số công nghệ sắp xếp trình tự hiện đại khác nhau mà cách mạng hóa các nghiên cứu về gen và sinh học phân tử. Những kỹ thuật này là trình tự Illumina, trình tự Roche 454, trình tự trình tự Ion Proton và trình tự của SOLiD (trình tự bởi Oligo Ligation Detection). Các hệ thống NGS nhanh hơn và rẻ hơn. Bốn phương pháp trình tự DNA chính được sử dụng trong các hệ thống NGS; pyrosequencing, trình tự bằng tổng hợp, sắp xếp bởi ligation và trình tự bán dẫn ion. Một số lượng lớn các chuỗi ADN hoặc RNA (hàng triệu) có thể được sắp xếp song song. Nó cho phép sắp xếp toàn bộ bộ gen của sinh vật trong một thời gian ngắn, không giống như trình tự Sanger mà mất nhiều thời gian hơn.
NGS có nhiều ưu điểm so với phương pháp sắp xếp truyền thống của Sanger. Đây là quy trình tốc độ cao, chính xác và hiệu quả hơn, có thể được thực hiện với quy mô mẫu nhỏ. NGS có thể được sử dụng trong nghiên cứu metagenomic, trong việc phát hiện các biến thể trong một genome cá nhân do chèn và xóa, vv và trong việc phân tích các biểu hiện gen.

Hình 1: Sự phát triển trong NGS Sequencing
Trình tự sắp xếp của Sanger là gì?
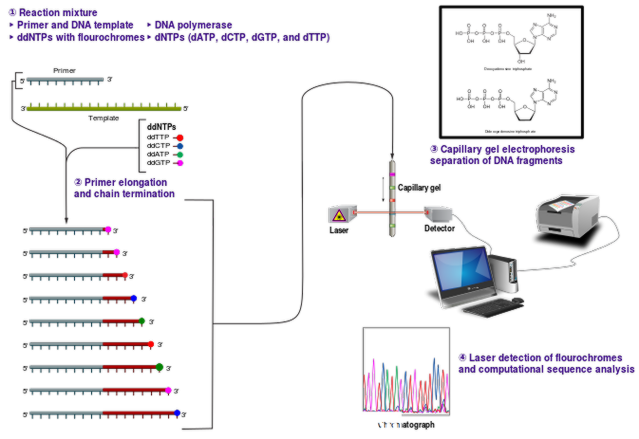
Sanger Sequencing là một phương pháp lập trình được phát triển bởi Frederick Sanger và các cộng sự của ông vào năm 1977 để xác định thứ tự nucleotide chính xác của một đoạn ADN nhất định. Nó còn được gọi là chuỗi kết thúc trình tự hoặc Dideoxy trình tự . Nguyên tắc làm việc của phương pháp này là việc chấm dứt quá trình tổng hợp sợi bằng cách kết hợp có chọn lọc của một chuỗi chấm dứt dideoxynucleotides (ddNTPs) như ddGTP, ddCTP, ddATP và ddTTP bởi DNA polymerase trong sự sao chép của DNA. Các nucleotide bình thường có 3 'nhóm OH để hình thành một liên kết phosphodiester giữa các nucleotides liền kề để tiếp tục sự hình thành sợi. Tuy nhiên, ddNTPs thiếu nhóm 3 'OH này và không thể tạo thành liên kết phosphodiester giữa các nucleotide. Do đó, sự kéo dài dây xích bị ngưng.
Trong phương pháp này, DNA đơn sợi phải được sắp xếp làm chuỗi mẫu cho tổng hợp DNA in vitro. Các yêu cầu khác là primer oligonucleotide, các tiền thân deoxynucleotide và enzyme DNA polymerase. Khi sự tiếp cận của đoạn cuối đã được biết đến, các mồi có thể dễ dàng được thiết kế để nhân bản DNA. Bốn phản ứng tổng hợp DNA được thực hiện trong bốn ống riêng biệt. Mỗi ống có ddNTPs riêng biệt cùng với các yêu cầu khác. Từ nucleotide cụ thể, một hỗn hợp của dNTPs và ddNTPs được thêm vào. Tương tự như vậy, bốn phản ứng riêng biệt được thực hiện trong bốn ống với bốn hỗn hợp. Sau các phản ứng, việc phát hiện các đoạn DNA và chuyển đổi mẫu phân đoạn thành thông tin trình tự được thực hiện. Kết quả các mảnh DNA bị phân hủy bởi nhiệt và tách ra bằng điện di gel. Nếu sử dụng các nucleotide phóng xạ, mô hình dải trong gel polyacrylamide có thể được hình dung bằng phép tự động. Khi phương pháp này sử dụng dideoxynucleotides có gắn huỳnh quang, nó có thể được giảm nhẹ xuống gel đọc và đi qua một chùm tia laser được phát hiện bởi máy dò huỳnh quang. Để tránh lỗi có thể phát sinh khi một chuỗi được đọc bởi mắt và nhập một cách thủ công vào máy tính, phương pháp này phát triển thành việc sử dụng tự động sequencer kết hợp với máy tính. Đây là phương pháp được sử dụng để tuần tự DNA từ dự án Hệ gen của con người. Phương pháp này vẫn đang được sử dụng với các sửa đổi tiên tiến vì nó cung cấp thông tin trình tự chính xác mặc dù là một quá trình tốn kém và chậm.
Hình 2: Sanger Sequencing
Sự khác nhau giữa NGS và Sanger Sequencing là gì?
- diff Điều Trung trước khi Bảng ->
NGS vs Sanger Sequencing
Next Generation Sequencing (NGS) đề cập đến quá trình trình tự thông lượng cao hiện đại.Nó mô tả một số công nghệ sắp xếp trình tự hiện đại khác nhau |
|
| Sanger Sequencing là một phương pháp lập trình được phát triển bởi Frederick Sanger để xác định trình tự nucleotide chính xác của một đoạn DNA nhất định. | Chi phí hiệu quả |
| NGS là một quy trình rẻ hơn vì nó làm giảm thời gian, sức mạnh con người và hóa chất. | |
| Đây là một quá trình tốn kém vì phải mất thời gian, sức mạnh của con người và nhiều hóa chất hơn. | Tốc độ |
| Nhanh hơn vì cả phát hiện hóa học và phát hiện tín hiệu của nhiều sợi đang diễn ra song song. | |
| Điều này tốn nhiều thời gian vì việc phát hiện và phát hiện hóa chất xảy ra theo hai quy trình riêng biệt và chỉ có thể đọc từng sợi một lần. | Độ tin cậy |
| NGS là đáng tin cậy. | |
| Trình tự Sanger có độ tin cậy | Kích cỡ mẫu |
| NGS ít đòi hỏi lượng DNA ít hơn. | |
| Phương pháp này cần một lượng lớn mẫu DNA. | Các căn cứ DNA trên mỗi đoạn tách rời |
| Số lượng cơ sở DNA trên một đoạn trình tự thấp hơn phương pháp Sanger | |
| Các trình tự tạo ra dài hơn các trình tự NGS. | Tóm lược - NGS và Sanger Sequencing |
NGS và Sanger Sequencing là các kỹ thuật sắp xếp trình tự nucleotide được sử dụng rộng rãi trong Sinh học phân tử. Trình tự Sanger là một phương pháp sắp xếp sớm đã được thay thế bằng NGS. Sự khác biệt chính giữa NGS và Sanger Sequencing là NGS là một quá trình tốc độ cao, chính xác và hiệu quả hơn so với trình tự Sanger. Cả hai kỹ thuật tạo ra các vụ bùng phát lớn trong Di truyền học và Công nghệ sinh học.
Tài liệu tham khảo:
1. Nowrousian, Minou. "Kỹ thuật tuần tự thế hệ tiếp theo cho các vi sinh vật tuyến sinh học: Các giải pháp dựa trên trình tự để giải quyết các vấn đề sinh học. " Tế bào nhân chuẩn. Hiệp hội vi trùng học Hoa Kỳ, tháng 9 năm 2010. Web. 18 tháng 2 năm 2017
2. Sanger, F., S. Nicklen và A. R. Coulson. "Trình tự DNA với các chất ức chế chấm dứt chuỗi. "Thủ tục của Học viện Khoa học Quốc gia 74. 12 (1977): 5463-467. Web.
3. Liu, Lin, Yinhu Li, Siliang Li, Ni Hu, Yimin He, Ray Pong, Danni Lin, Lihua Lu và Maggie Law. "So sánh các hệ thống trình tự thế hệ tiếp theo. "Tạp chí Khoa học Sinh học và Công nghệ sinh học 2012 (2012): 1-11. Web.
Hình ảnh:
"Sanger-sequencing" Tác giả Estevezj - Tác phẩm của chính mình (CC BY-SA 3. 0) thông qua Commons Wikimedia
"Những phát triển trong trình tự thế hệ kế tiếp" By Nederbragt, Lex (2012) - (CC BY 3. 0) thông qua Commons Wikimedia



